まず、各定数の確認する。
import numpy as np
elementary_charge = 1.602176634*10**-19
c_light_m = 299792458
electron_mass_kg = 9.1093837015*10**-31
permittivity_vacuum = 8.8541878128*10**-12
r_e = 2.8179403262*10**-13
print("r_e =", elementary_charge**2 / (4 * np.pi * permittivity_vacuum * electron_mass_kg * c_light_m**2)*100,"cm")
electron_mass = 0.510998928
Na = 6.02214179*10**23
dedx_constant = 0.3070749187
print("dedx_constant =", 4 * np.pi * Na * electron_mass * r_e**2,"MeV/cm2")
色んな項を無視したエネルギー損失と、エネルギーストラグリングを計算する。
def energy_loss_straggling(p_A, p_Z, p_Q, p_T, t_Z, shell_correction = False, LSX=1):
'''
p_A=238 # projectileのA
p_Z=92 # projectileのZ
p_Q=92 # projectileのQ
p_T = 345 #projectileのEnergy MeV/u
t_Z = 4 # tagetのZ
'''
import numpy as np
electron_mass = 0.510998928
Na = 6.02214179*10**23
dedx_constant = 0.3070749187
atomic_mass_unit = 931.4940954
zp_eff = p_Q
gamma=1.0 + p_T/atomic_mass_unit
beta2=1.0-1.0/(gamma*gamma)
beta = np.sqrt(beta2)
def get_a(t_Z):
standard_weights = [1.008, 4.003,\
6.941, 9.012, 10.81, 12.01, 14.01, 16.00, 19.00, 20.18,\
22.99, 24.31, 26.98, 28.09, 30.97, 32.07, 35.45, 39.95,\
39.10, 40.08, 44.96, 47.87, 50.94, 52.00, 54.94, 55.85, 58.93, 58.69, 63.55, 65.38, 69.72, 72.63, 74.92, 78.97, 79.90, 83.80,\
85.47, 87.62, 88.91, 91.22, 92.91, 95.95, 99, 101.1, 102.9, 106.4, 107.9, 112.4, 114.8, 118.7, 121.8, 127.6, 126.9, 131.3,\
132.9, 137.3,\
138.9, 140.1, 140.9, 144.2, 145, 150.4, 152.0, 157.3, 158.9, 162.5, 164.9, 167.3, 168.9, 173.0, 175.0,\
178.5, 180.9, 183.8, 186.2, 190.2, 192.2, 195.1, 197.0, 200.6, 204.4, 207.2, 209.0, 210, 210, 222,\
223, 226,\
227, 232.0, 231.0, 238.0, 237, 239, 243, 247, 247, 252, 252, 257, 258, 259, 262,\
267, 268, 271, 272, 277, 276, 281, 280, 285, 278, 289, 289, 293, 293, 294]
return standard_weights[t_Z-1]
t_A = get_a(t_Z)
def get_ipot(t_Z):
arr_ipot = [
19.2, 41.8, \
40.0, 63.7, 76.0, 81.0, 82.0, 95.0, 115.0,137.0,\
149.0,156.0,166.0,173.0,173.0,180.0,174.0,188.0,\
190.0,191.0,216.0,233.0,245.0,257.0,272.0,286.0,297.0,311.0,322.0,330.0,334.0,350.0,347.0,348.0,343.0,352.0,\
363.0,366.0,379.0,393.0,417.0,424.0,428.0,441.0,449.0,470.0,470.0,469.0,488.0,488.0,487.0,485.0,491.0,482.0,\
488.0,491.0,501.0,523.0,535.0,546.0,560.0,574.0,580.0,591.0,614.0,628.0,650.0,658.0,674.0,684.0,694.0,705.0,718.0,727.0,736.0,746.0,757.0,790.0,790.0,800.0,810.0,823.0,823.0,830.0,825.0,794.0,\
827.0,826.0,841.0,847.0,878.0,890.0,900.0,910.0,920.0,930.0,940.0,950.0,960.0,970.0,980.0,990.0,1000.,1010.,1020.,1030.,1040.,1050.,1060.,1070.,1080.,1090.,1100.,1110.,1120.,1130.,1140.,1150.
]
return arr_ipot[t_Z-1]
Ipot = get_ipot(t_Z)
f1 = dedx_constant*pow(zp_eff,2.0)*t_Z/(beta2*t_A)
f2 = np.log(2.0*electron_mass*beta2*gamma*gamma/(Ipot/1000000)) - beta2
cor = 0
f2 = f2 - cor/t_Z
barkas = 1.0
LS = 0.0
delta = 0
energy_loss = ((f2)*barkas + LS - delta/2.)*f1
energy_straggling = f1 * electron_mass * beta2 * gamma * gamma * LSX
return energy_loss, energy_straggling
dedxの定数 dedx_constant=Kを次式のように定義すると、
諸々の補正項を無視するとdE/dxは次式のように表現される。
energy loss straggling Ωは次式のように表現される。
345MeV/uの238U、320MeV/uの4Heを、ベリリウムの薄膜に入射したした場合の計算。
print(energy_loss_straggling(p_A=238, p_Z=92, p_Q=92, p_T=345, t_Z=4))
print(energy_loss_straggling(p_A=4, p_Z=2, p_Q=2, p_T=320, t_Z=4))
(22419.411135356077, 1107.0218819306313)
(11.02602078859991, 0.5028755625661142)
と返ってくるはずである。
標的をBe1mmとすると、物質量0.185 g/cm2をかければ、Energy loss と stragglingの2乗の値が得られる。345MeV/uの238U92+ビームのEnergy lossは 4147.6 MeV、Energy stragglingは14.3 MeVとなる。それぞれAで割ると、Energy lossは17.43±0.06 MeV/uとなる。LISE++で出力される値は17.85±0.08 MeV/uである。
320MeV の 4HeビームがBe標的1mmに入射したとき、Energy lossは2.04 MeV、Energy stragglingは0.31 MeVである。それぞれAで割ると、Energy lossは0.510±0.076 MeV/uとなる。LISE++の値は0.508±0.068 MeV/uである。
精度よくEnergy lossを求めるためには、barkas項、delta項、LS項を与える必要がある。
stragglingにかけるべきXの値はATIMAで計算でき、それを与えるとLISE++の値とほぼ一致する。
よくあるStopping powerの図を描く。粒子は陽子、標的は銅(Z=29)にして、横軸はβγにした。
import matplotlib.pyplot as plt
import numpy as np
elosses = []
betagammas = []
for log_energy in range(-10,50):
energy = 10**(log_energy*0.1)
eloss, strag = energy_loss_straggling(p_A=1, p_Z=1, p_Q=1, p_T=energy, t_Z=29)
atomic_mass_unit = 931.4940954
gamma=1.0 + energy/atomic_mass_unit
beta2=1.0-1.0/(gamma*gamma)
beta = np.sqrt(beta2)
betagammas.append(beta*gamma)
elosses.append(eloss)
plt.rcParams["font.size"] = 14
plt.rcParams['figure.figsize'] = [7, 5]
plt.plot(betagammas,elosses)
plt.xscale("log")
plt.yscale("log")
plt.ylim(1,None)
plt.xlabel("βγ")
plt.ylabel("MeV / (g/cm2)")
plt.show()

PDGの2018年版ブックレットの図33.1と比較する。
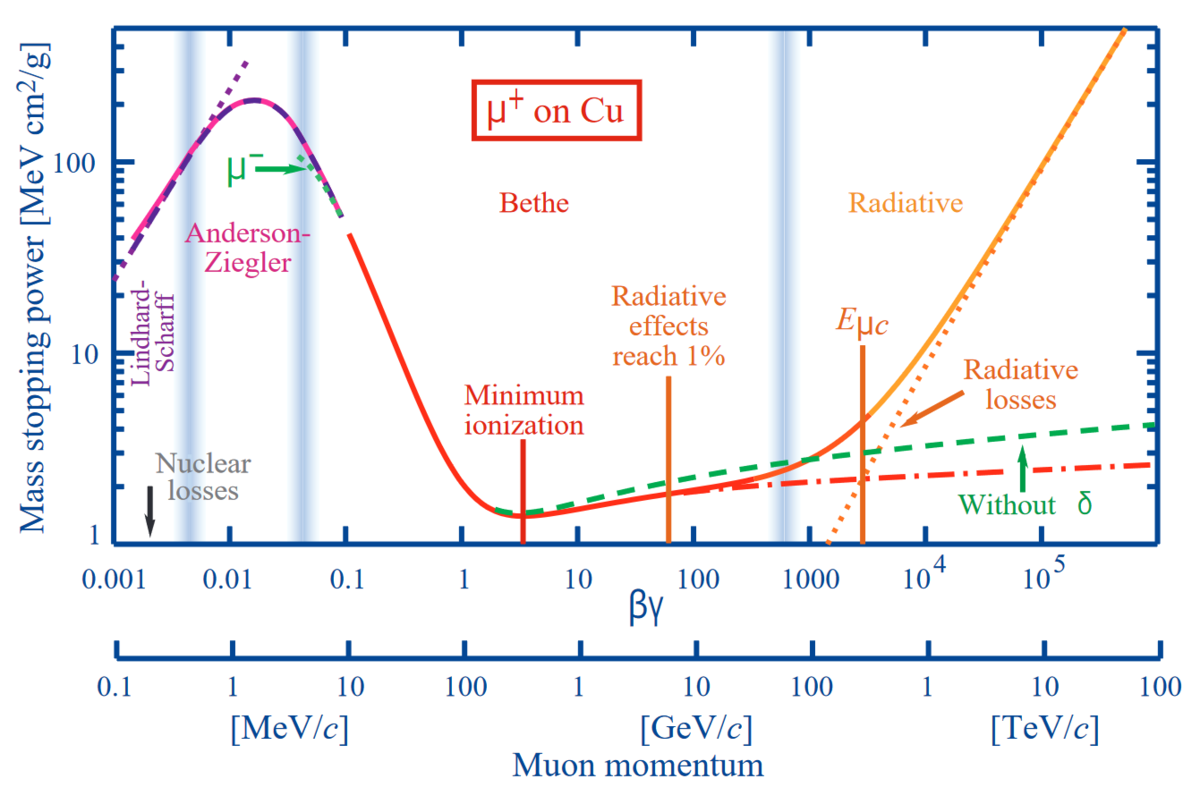
概ね問題なさそうだ。
密度補正deltaの部分は比較的簡単にPythonで書けた
def bethek_density_effect(beta, t_Z, beta, gamma):
'''
密度効果補正
'''
del_0= [
0.0, 0.00, 0.14, 0.14, 0.14, 0.12, 0.00, 0.00, 0.00, 0.00,
0.08, 0.08, 0.12, 0.14, 0.14, 0.14, 0.00, 0.00, 0.10, 0.14,
0.10, 0.12, 0.14, 0.14, 0.14, 0.12, 0.12, 0.10, 0.08, 0.08,
0.14, 0.14, 0.08, 0.10, 0.00, 0.00, 0.14, 0.14, 0.14, 0.14,
0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14,
0.14, 0.14, 0.00, 0.00, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14,
0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14,
0.14, 0.14, 0.14, 0.14, 0.08, 0.10, 0.10, 0.12, 0.14, 0.14,
0.14, 0.14, 0.14, 0.14, 0.00, 0.00, 0.00, 0.14, 0.14, 0.14,
0.14, 0.14, 0.14, 0.14, 0.14, 0.14, 0.14 ]
x0= [
1.8639, 2.2017, 0.1304, 0.0592, 0.0305, -.0178, 1.7378, 1.7541, 1.8433, 2.0735,
0.2880, 0.1499, 0.1708, 0.2014, 0.1696, 0.1580, 1.5555, 1.7635, 0.3851, 0.3228,
0.1640, 0.0957, 0.0691, 0.0340, 0.0447, -.0012, -.0187, -.0566, -.0254, 0.0049,
0.2267, 0.3376, 0.1767, 0.2258, 1.5262, 1.7158, 0.5737, 0.4585, 0.3608, 0.2957,
0.1785, 0.2267, 0.0949, 0.0599, 0.0576, 0.0563, 0.0657, 0.1281, 0.2406, 0.2879,
0.3189, 0.3296, 0.0549, 1.5630, 0.5473, 0.4190, 0.3161, 0.2713, 0.2333, 0.1984,
0.1627, 0.1520, 0.1888, 0.1058, 0.0947, 0.0822, 0.0761, 0.0648, 0.0812, 0.1199,
0.1560, 0.1965, 0.2117, 0.2167, 0.0559, 0.0891, 0.0819, 0.1484, 0.2021, 0.2756,
0.3491, 0.3776, 0.4152, 0.4267, 0.4300, 1.5368, 0.6000, 0.5991, 0.4559, 0.4202,
0.3144, 0.2260, 0.1869, 0.1557, 0.2274, 0.2484, 0.2378]
gamma = 1/np.sqrt(1-(beta*beta))
x = np.log(beta * gamma) / 2.3025851
delta = del_0[t_Z-1] * pow(10.0,(2.*(x-x0[t_Z-1])))
return delta
全てのコードのまとめ
gitlab.com
参照: C++のCATIMAコード






